Akoya Biosciences空間生物學分析平台簡介
Spatial Biology 空間生物學係藉由組織空間內的細胞組成、細胞分佈與功能性生物分子來定義功能性區域,並了解細胞族群的互動關係。以癌症生物學為例,免疫腫瘤微環境(iTME)牽涉非常多種免疫細胞與非免疫細胞,要了解這些群集細胞聚集的地方以及互動關聯,需要whole slide以及single cell等級的大數據影像資料才能進行完整的分析。
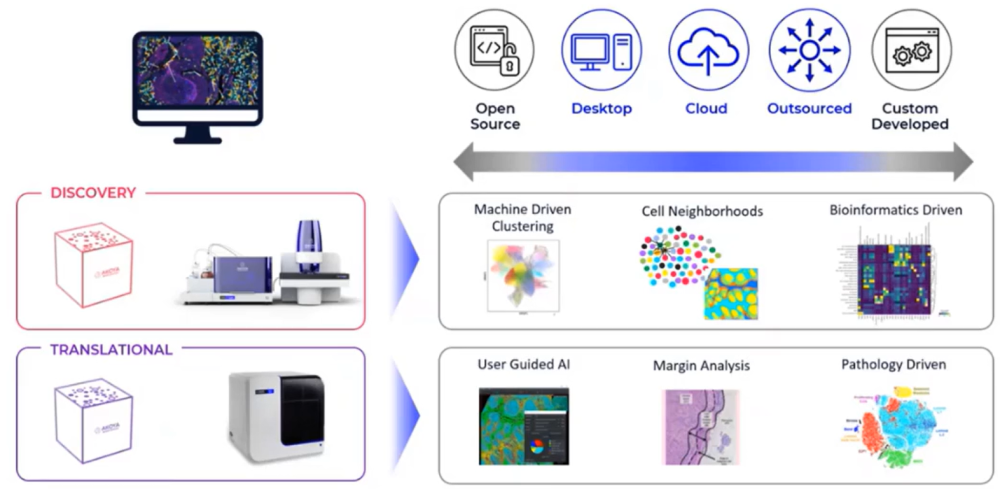
然而,現今空間生物學領域主要的阻礙在於影像平台的產出速度、複雜的實驗流程及巨量的資料不利於儲存及傳輸,因而限制整體通量。為了克服這些困難,Akoya Biosciences利用QPTIFF檔案格式作為組織影像空間分析方案的核心。此種檔案結合影像處理演算法,保留高品質空間影像並增加分析流程的效率。
QPTIFF影像檔案可進入inForm分析軟體進行光譜拆解(spectral unmixing),分離不同marker的螢光訊號,接著進行細胞分割(cell segmentation)、組織辨認(tissue segmentation)及細胞分型(cell phenotyping)或細胞評分(scoring)等分析項目。將訓練軟體後的方法儲存,可以進一步套用到其他影像實現組織分析自動化。
Akoya分析方法不限於自家的分析軟體,QPTIFF檔案也可以輸入QuPath分析軟體進行細胞分割並進一步訓練細胞分型,辨認不同表型的細胞。在QuPath輸出細胞空間資訊以及marker表現資料後,還可以進一步輸入到CytoMAP分析軟體進行分析,利用演算法將細胞群集(cell clustering)接著進一步進行細胞鄰域空間分析(Cellular neighborhoods spatial map)或細胞鄰域互動關係分析(Region interactions)等。
Phenochart數位切片看圖軟體 
Phenochart是一款能觀看組織數位切片影像以及註記分析區域的軟體。在PhenoImager Fusion以及PhenoImager HT 拍攝組織影像之後,即可在Phenochart立即觀看拍攝結果,並可進行光譜拆解去除自體螢光並輸出影像。在註記分析區域之後,檔案即可進一步在inForm軟體進行組織,細胞的分析。

(PhenoImager系列拍攝多重螢光影像之後將檔案輸入phenochart軟體,可查看結果、光譜拆解或標記分析區域)
分析軟體介紹
1. inForm進階影像分析軟體: ![]()
1.1 軟體簡介
inForm 為Akoya專利影像分析軟體,具有機器學習功能,可進行光譜拆解、組織辨認、細胞辨認、細胞分群及細胞空間解析,定性、定量、螢光colocalization 皆可計算。使用者可訓練軟體演算法去辨認組織型態,再將訓練結果套用到所有的組織影像即可批次自動辨認組織型態或細胞表型。軟體除可以讀取QPTIFF及多光譜影像.im3之外,亦可讀取JPEG, single-layer TIFF, BMP及PNG檔案。完成分析後也可將資訊進一步輸出到PhenoptrReports進行細胞距離運算分析。
1.2 inForm的重點功能與特色:
- 一站式分析流程

在開始分析影像之前,使用者可在這個一站式分析流程架構下選擇自己想要的流程。決定分析架構之後,由左至右完成所有分析及結果輸出。
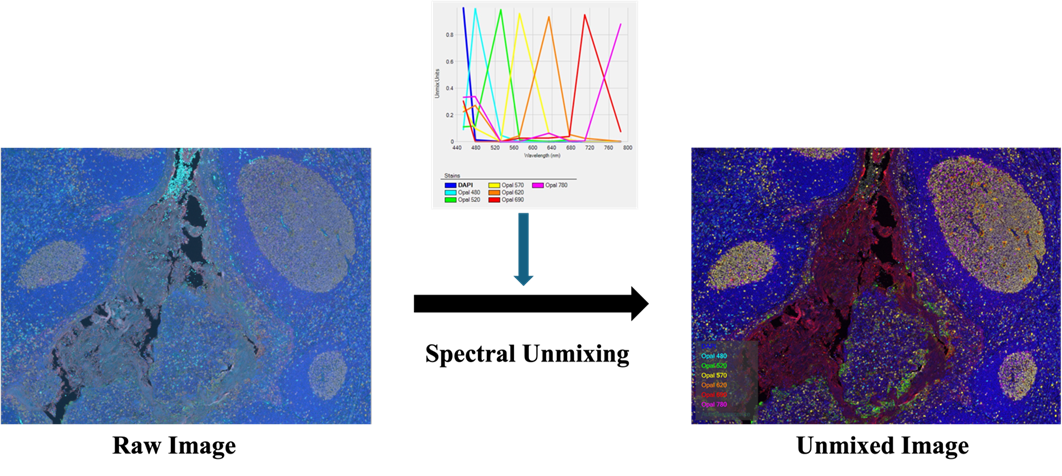
- Spectral Unmixing光譜拆解
將軟體內建的染劑資料庫套用至影像進行光譜拆解,去除自體螢光訊號同時免除不同訊號間的干擾。
- Tissue segmentation組織劃分
如下圖,標記tumor以及stroma組織區域並配合特定marker訊號來訓練軟體,最後由軟體分割全區組織影像。

- Cell segmentation細胞劃分
定義細胞參數進行細胞劃分。
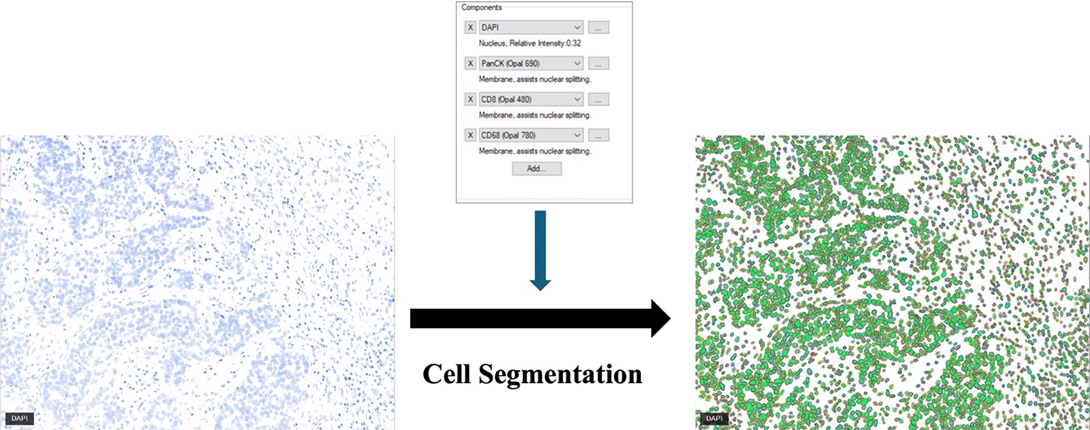
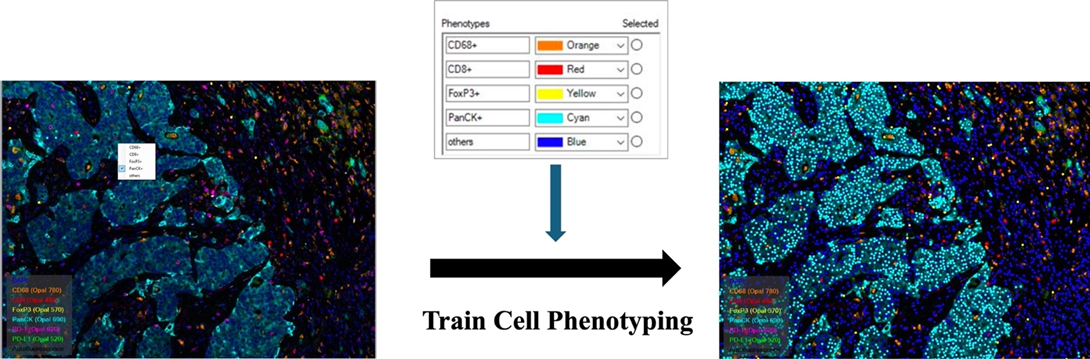
- Cell Phenotyping細胞分型
自行對軟體定義細胞型態CD68+/CD8+/ FoxP3+/ PanCK+/others並針對每個表型標記數個細胞,接著訓練軟體對全影像的細胞分型。
- IHC/IF Scoring定量評分
如下圖,以4-bin為例,將方法設定為4區評分。選擇定量目標為PD-L1並設定評分門檻進行定量評分比較。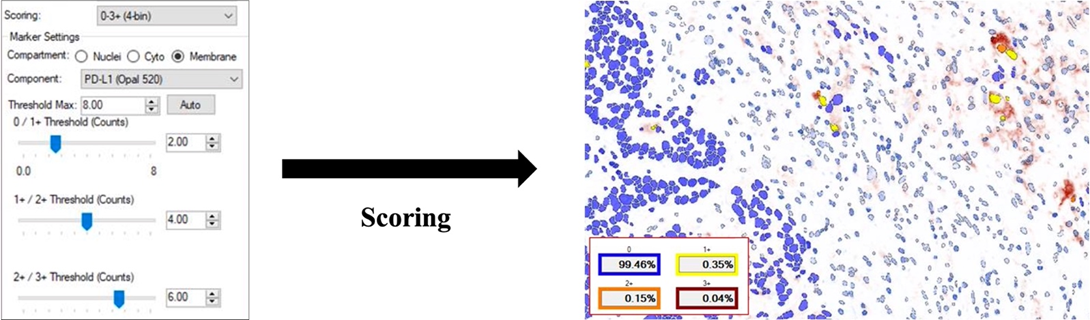
1.3軟體分析流程
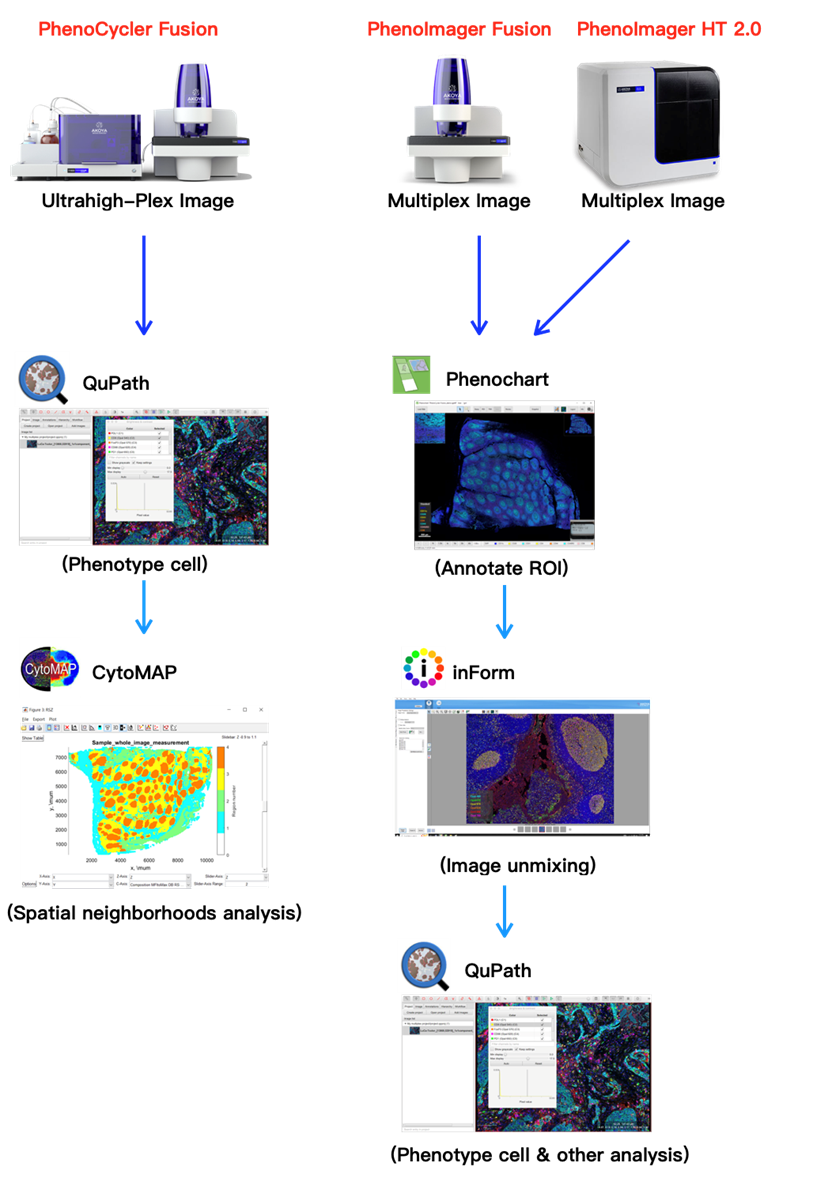
從Phenochart看圖軟體開始進行分析區域的標記,並接著輸入inForm進行組織辨識、細胞分割、細胞分型、定量評分等分析。若要運算細胞間距離資訊可從inForm輸出結果到PhenoptrReports進一步運算分析。
2. QuPath數位病理/生物影像分析軟體:
2.1 軟體簡介
QuPath為一種生物影像開源軟體,主要針對數位病理領域whole slide影像進行分析。與其他分析軟體最大的不同在於QuPath為階層結構以及object-based的data model分析。軟體支援多種影像格式如Bio-Formats以及OpenSlide的全片影像,其功能涵蓋multiplexed影像進行觀看、註記,細胞biomarker定量、自動化細胞劃分(Cell segmentation)以及細胞空間定型(Spatial phenotyping)。為了精確在組織影像上劃分細胞,Akoya提供PhenoCycler系列客戶客製的 StarDist物件偵測語法,適用於多數組織。QuPath 也具備機器學系功能,使用者可藉由標記特定表型的細胞來訓練軟體,最後將分類器套用到整張影像完成細胞分型。完成細胞分割及細胞分型後,QuPath可輸出細胞的marker以及XY座標資訊,並將結果導入CytoMAP軟體,進一步分析細胞鄰域、群集互動關係。
2.2軟體功能與特色
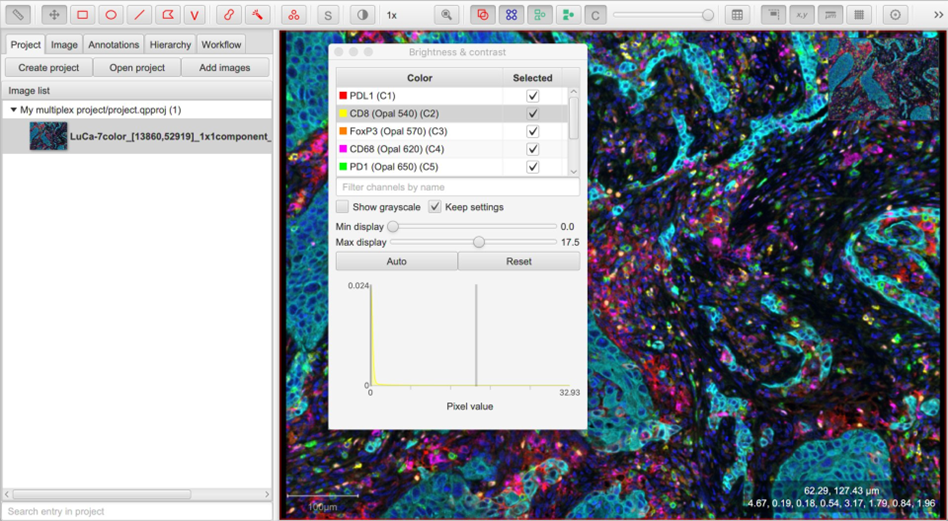
- Visualization 影像觀看: 設定每個channel的名稱及顏色,可選擇性呈現特定marker顯色。
- Cell segmentation細胞劃分
插入Akoya 訓練的StarDist模型,定出每顆細胞區域。
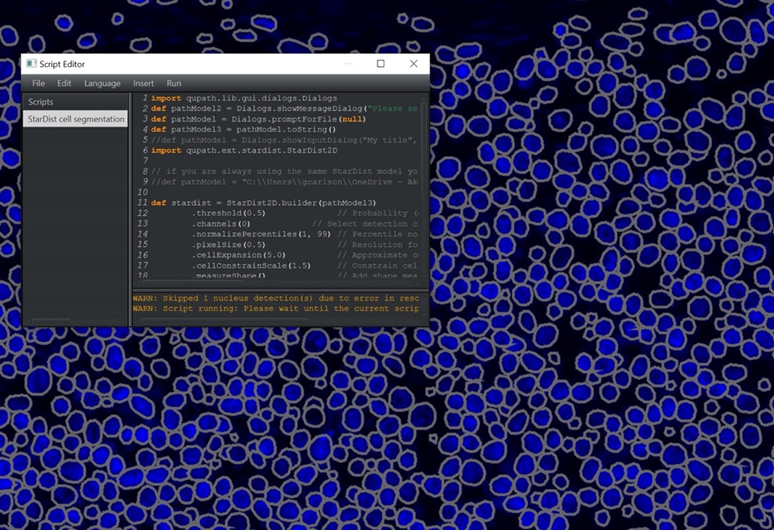
- Phenotyping細胞分型
標記特定細胞型來訓練分類器,完成訓練後可將分類器用於整張組織影像或其他影像完成細胞分型。
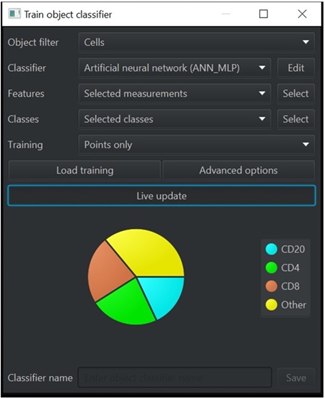

2.3 軟體分析流程
PCF產出的ultrahigh-plex影像可直接輸入QuPath進行細胞分割、細胞分型等分析,並進一步輸出結果至CytoMAP進行細胞群集、細胞鄰域分析。Fusion或PI HT的multiplex影像由Phenochart進行區域註記再送入inForm進行光譜拆解,分離訊號之後接著輸入到QuPath進行細胞分型等分析。

3. CytoMAP組織細胞多維度分析軟體:
3.1 軟體簡介
CytoMAP是一種組織細胞多維度分析開源軟體,主要針對含有位置資訊的single-cell資料進行分析。其分析方法是透過神經網路為主體的clustering演算法進行運算,並把這些演算法清楚地整合在軟體裡面,介面簡潔操作容易。軟體可輸入的資料種類包含.csv、.wsp+.fcs及.mat等,主要功能為空間上的細胞鄰域分析(Cellular neighborhood analysis)、鄰域互動關係(Region interactions)及降維(Dimension reduction)群集分析等等。
3.2 軟體功能與特色
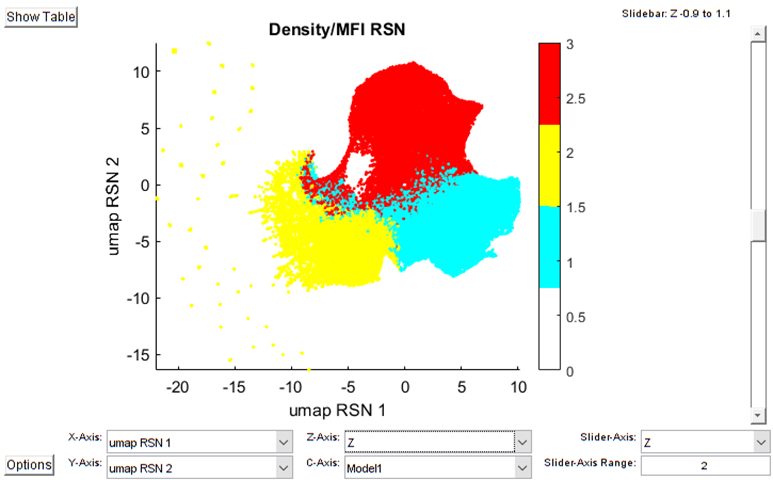
- Cellular neighborhoods spatial map細胞鄰域空間圖 : 定義Neighborhoods之後,將Neighborhoods群集為regions產生細胞鄰域空間圖。
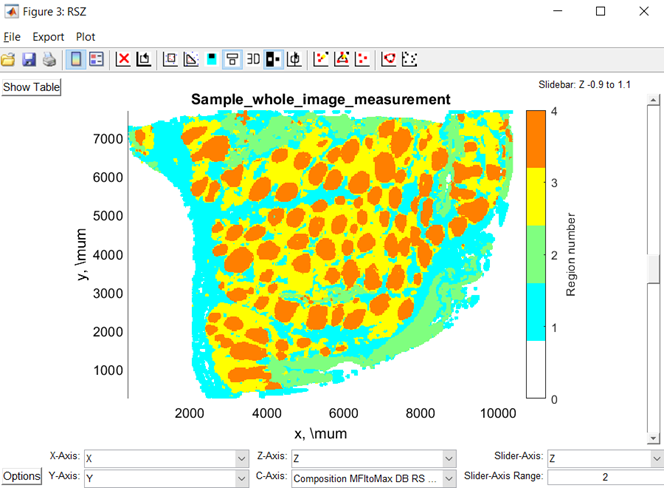
- Region interactions鄰域互動關係 : 計算不同細胞鄰域邊界相鄰的周長,呈現細胞鄰域互動關係。
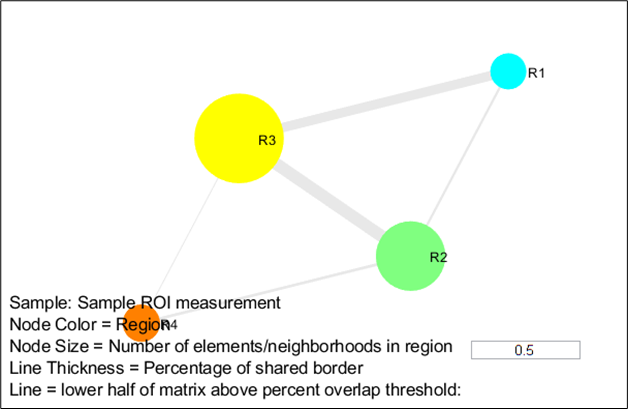
- Dimension reduction細胞鄰域降維分析 : 選擇想用的降維度演算法如t-SNE或UMAP將所有細胞鄰域的分群結果以2D投影方式簡化資料呈現於圖表。
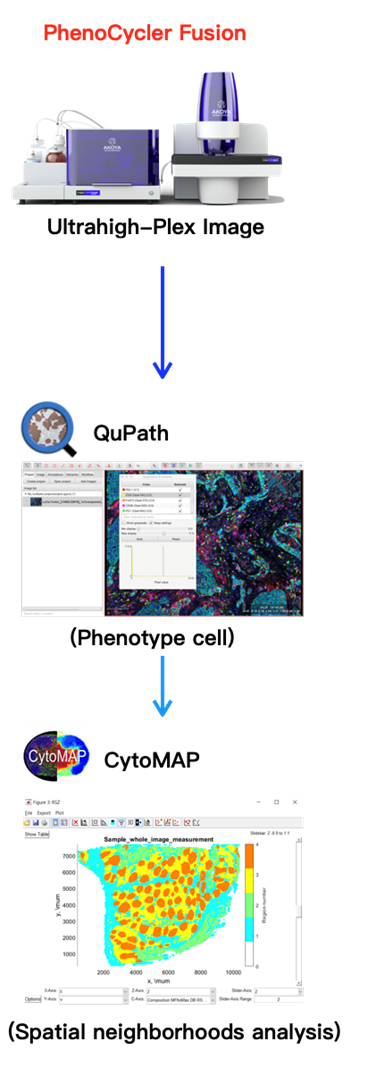
 3.3 軟體分析流程
3.3 軟體分析流程
PCF產出的ultrahigh-plex影像可直接輸入QuPath進行細胞分割、細胞分型等分析,並進一步輸出結果至CytoMAP進行細胞群集、細胞鄰域分析。
英文說明
inForm:
inForm® projects can be tailored to enable biomarker analysis in both solid tissues and TMAs and handle a variety of stains (H&E, multiplexed IHC, and multiplexed immunofluorescence). Its powerful unmixing engine, enables the separation and measurement of weak and spectrally overlapping markers in multiplex assays. Automated, trainable algorithms permit detection, cell segmentation, tissue segmentation and identification of multiple markers within a sample. Once trained, inForm will locate and analyze user-specified regions automatically across an entire image or multiple images. Large numbers of images can be rapidly batch processed, allowing analysis that might have taken days to be done in a matter of minutes.
QuPath:
QuPath is open-source software for bioimage analysis and is often used for digital pathology applications that offer a powerful set of tools for working with whole slide images. It offers an array of novel algorithms to provide not only ready-made, user-friendly solutions to common, challenging analysis problems in pathology, but also the building blocks to create custom workflows.
CytoMAP:
CytoMAP is intended to make advanced data analytic techniques accessible for single-cell data with position information. The goal is to take established analytical techniques, such as neural network based clustering algorithms, and package them in a user friendly way, allowing researchers to use them to explore complex data sets.